Ejemplo con Datos demográficos#
#!git clone https://github.com/cgg-upm/upm-dma-som
#!dir upm-dma-som\som
#!copy upm-dma-som\som\som.py .\*.*
#!copy upm-dma-som\som\utils.py .\*.*
from som import som
from utils import somutils
import numpy as np
import pandas as pd
Ejemplo datos Población de Irlanda#
Datos: censo irlandés, area de Dublín, año 2011
Visualización de los resultados
Los datos cocinados en : dataIrelandPopulationSOM.csv
df = pd.read_csv('data/dataIrelandPopulationSOM.csv')
df.head()
| Unnamed: 0 | id | avr_age | avr_household_size | avr_education_level | avr_num_cars | avr_health | rented_percent | unemployment_percent | internet_percent | single_percent | married_percent | separated_percent | divorced_percent | widow_percent | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 15687 | 267123023 | 40.028112 | 2.524752 | 3.038462 | 1.039604 | 4.385542 | 6.930693 | 15.343915 | 71.000000 | 53.413655 | 33.333333 | 4.819277 | 2.811245 | 5.622490 |
| 1 | 13895 | 267016001 | 35.673660 | 3.320611 | 3.597701 | 1.983740 | 4.509434 | 4.878049 | 12.461059 | 72.950820 | 49.417249 | 44.988345 | 1.398601 | 0.233100 | 3.962704 |
| 2 | 13896 | 267016002 | 35.882353 | 3.324324 | 4.295302 | 1.905405 | 4.596639 | 1.351351 | 10.404624 | 83.783784 | 47.478992 | 43.697479 | 3.361345 | 0.000000 | 5.462185 |
| 3 | 13729 | 267002034 | 38.516667 | 3.088608 | 3.871795 | 1.730769 | 4.530172 | 3.896104 | 8.108108 | 78.947368 | 47.083333 | 48.333333 | 0.833333 | 1.250000 | 2.500000 |
| 4 | 13724 | 267002029 | 24.678005 | 3.512000 | 3.933735 | 1.112000 | 4.510345 | 20.800000 | 21.810700 | 81.300813 | 67.120181 | 26.984127 | 2.267574 | 1.587302 | 2.040816 |
df.columns
Index(['Unnamed: 0', 'id', 'avr_age', 'avr_household_size',
'avr_education_level', 'avr_num_cars', 'avr_health', 'rented_percent',
'unemployment_percent', 'internet_percent', 'single_percent',
'married_percent', 'separated_percent', 'divorced_percent',
'widow_percent'],
dtype='object')
df.values.shape
(4806, 15)
columns=['avr_age','avr_education_level','avr_num_cars','unemployment_percent']
X = df[columns].to_numpy()
X.shape
(4806, 4)
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
X_std = scaler.fit_transform(X)
X_std.shape
(4806, 4)
Definir y entrenar el SOM#
nrows = 10
ncols = 10
#dimensions = X_std.shape[1]
dimensions = X.shape[1]
#vicinity = "rectangular"
vicinity = "hexagonal"
smm = som(nrows=nrows,
ncols=ncols,
dimension=dimensions,
vicinity=vicinity)
smm.train_SOM(train_data=X,
epochs=5)
Show code cell output
0%| | 0/5 [00:00<?, ?it/s]
20%|████████████████████████████▍ | 1/5 [00:00<00:03, 1.30it/s]
40%|████████████████████████████████████████████████████████▊ | 2/5 [00:01<00:02, 1.25it/s]
60%|█████████████████████████████████████████████████████████████████████████████████████▏ | 3/5 [00:02<00:01, 1.24it/s]
80%|█████████████████████████████████████████████████████████████████████████████████████████████████████████████████▌ | 4/5 [00:03<00:00, 1.23it/s]
100%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████| 5/5 [00:04<00:00, 1.19it/s]
100%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████| 5/5 [00:04<00:00, 1.22it/s]
<som.som at 0x1ade7bde5d0>
Gráfico con el clustering y el recuento de nodos#
somutils.plot_Cluster(pSom=smm, n_clusters=4, figsize=(7,5))
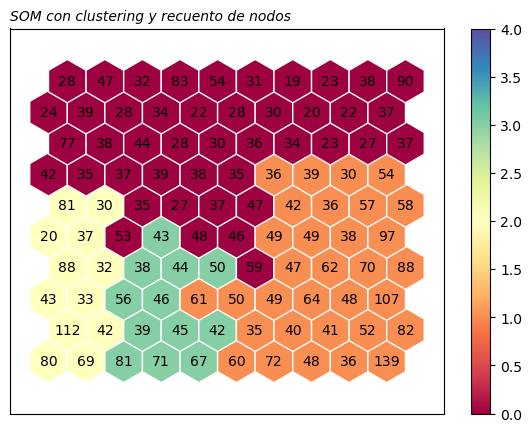
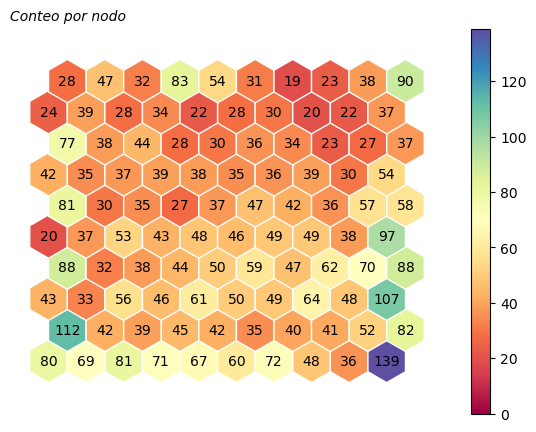
Recuento de observaciones por neurona#
somutils.plot_neurPointsCount(pSom=smm, figsize=(7,5))
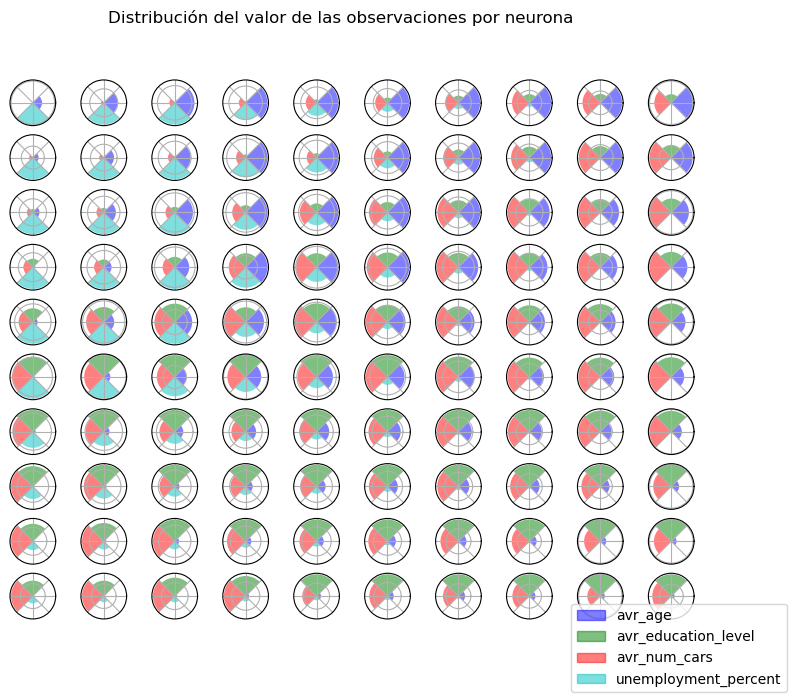
Mapeo de valores#
somutils.plot_valuesMap(pSom=smm, labels=columns, figsize=(9,7))
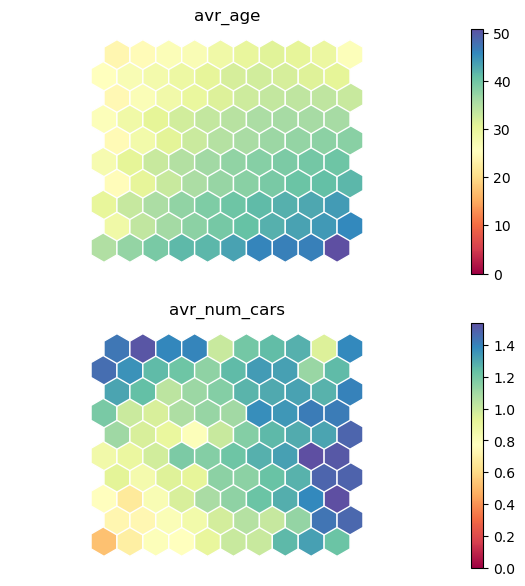
Mapas de calor#
somutils.plot_heatmaps(pSom=smm, labels=columns, ilabMap=[0], figsize=(7,5))

somutils.plot_heatmaps(pSom=smm, labels=columns, ilabMap=[0,2], figsize=(7,7))
Mapeo de observaciones sobre cada neurona#
somutils.plot_pointsMap(pSom=smm, figsize=(7,7))
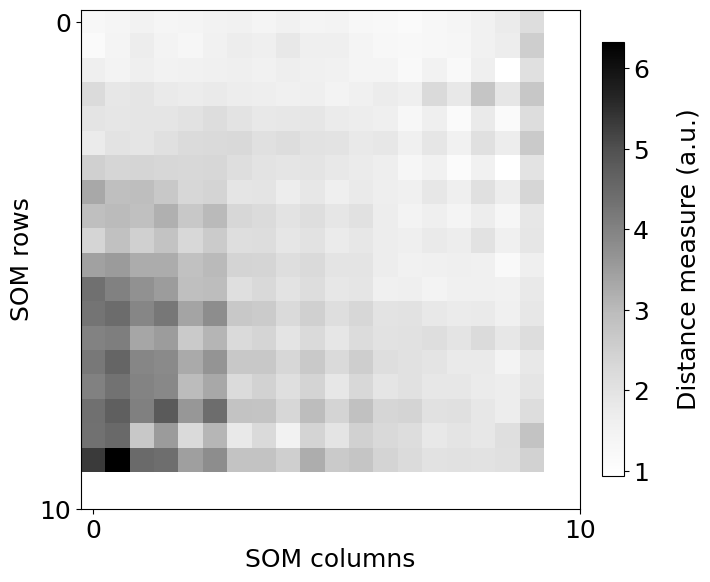
Dibujo de la matriz de distancias unificadas (U-Matrix)#
Contiene la distancia entre los nodos del SOM. En el gráfico a más intensidad hay una menor distancia entre las neuronas adyacentes. Para cada neurona se calcula la distancia entre ella y sus neuronas vecinas más próximas. Cuanto menor sea el resultado, más próxima estará la neurona con sus vecinas.
somutils.plot_u_matrix(pSom=smm, figsize=(7,7))
Distancia media a la neurona más próxima por iteración#
Acceder a los datos asociados a cada neurona#
Índices de las observaciones de una neurona, por ejemplo la neurona del nodo (0, 0)
Se muestra sólo la dimensión con len
len(smm.getIndexDataNeur(0, 0))
80
Y los datos de las observaciones asociados a la neurona (0,0)
Se muestra sólo la dimensión con shape
smm.getDataNeuron(0, 0).shape
(80, 4)
Clústeres sobre el mapa topográfico#
Si los datos contienen información GIS, es posible representar qué puntos del mapa topográfico de Dublín están relacionados con qué clústeres.
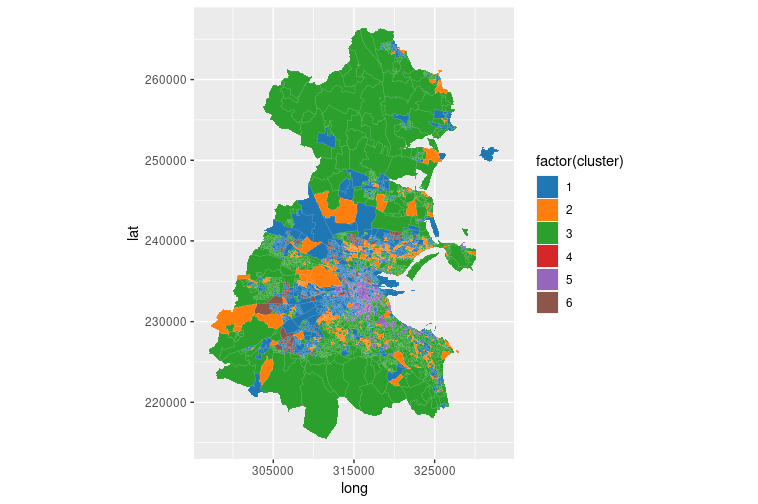
Fig. 37 Geolocalización de los clústeres hallados#
