Práctica 2. Continuación#
SOM como un Clasificador#
Los mapas auto-organizativos de Kohonen también se pueden utilizar como clasificadores mediante un aprendizaje supervisado que utiliza parte de los datos para entrenar la red y ajustar los pesos de cada neurona de manera que se minimize el error de salida y deja otra parte de los datos para comprobar si el entrenamiento ha sido eficaz.
Ejemplo 1. Selección de un conjunto de entrenamiento#
Elige de manera aleatoria un conjunto de entrenamiento que contenga un 70% de los datos de som.wine (manteniendo la primera columna que contiene la información de la bodega de origen). A partir de este conjunto, creamos el conjunto de entrenamiento y el conjunto de comprobación escalados.
Para que la comparación sea adecuada, el segundo se debe escalar con referencia al conjunto de entrenamiento. A partir de éstos, creamos dos listas donde detallamos el conjunto que se va comprobar respecto a que atributo, en nuestro caso, las bodegas (Cultivars)
Las bodegas son:
Cultivars grapes = (Nebbiolo, Barberas, Grignolino)
#!git clone https://github.com/cgg-upm/upm-dma-som
#!dir upm-dma-som\som
#!copy upm-dma-som\som\som.py .\*.*
#!copy upm-dma-som\som\utils.py .\*.*
from som import som
from utils import somutils
import numpy as np
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import ConfusionMatrixDisplay
from sklearn.metrics import confusion_matrix
# Se preparan las columnas a tratar
df_wine = pd.read_csv('data/wine.csv', header=None)
df_wine.columns = ['label', 'alcohol',
'malic acid', 'ash',
'ash alkalinity',
'magnesium', 'tot. phenols',
'flavanoids', 'non-flav. phenols',
'proanth',
'col. int.', 'col. hue',
'OD ratio'
,'proline']
# Se borra la columna de la etiqueta
#df_wine = df_wine.drop('label', axis=1)
df_wine.head()
| label | alcohol | malic acid | ash | ash alkalinity | magnesium | tot. phenols | flavanoids | non-flav. phenols | proanth | col. int. | col. hue | OD ratio | proline | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 14.23 | 1.71 | 2.43 | 15.6 | 127 | 2.80 | 3.06 | 0.28 | 2.29 | 5.64 | 1.04 | 3.92 | 1065 |
| 1 | 1 | 13.20 | 1.78 | 2.14 | 11.2 | 100 | 2.65 | 2.76 | 0.26 | 1.28 | 4.38 | 1.05 | 3.40 | 1050 |
| 2 | 1 | 13.16 | 2.36 | 2.67 | 18.6 | 101 | 2.80 | 3.24 | 0.30 | 2.81 | 5.68 | 1.03 | 3.17 | 1185 |
| 3 | 1 | 14.37 | 1.95 | 2.50 | 16.8 | 113 | 3.85 | 3.49 | 0.24 | 2.18 | 7.80 | 0.86 | 3.45 | 1480 |
| 4 | 1 | 13.24 | 2.59 | 2.87 | 21.0 | 118 | 2.80 | 2.69 | 0.39 | 1.82 | 4.32 | 1.04 | 2.93 | 735 |
X, y = df_wine.values[:,1:14], df_wine.values[:,0]
scaler = StandardScaler()
X_std = scaler.fit_transform(X)
X_train, X_test, y_train, y_test =train_test_split(X_std, y, test_size=0.3, random_state=1, stratify=y)
df = pd.DataFrame(X_train)
df.columns = ['alcohol',
'malic acid', 'ash',
'ash alkalinity',
'magnesium', 'tot. phenols',
'flavanoids', 'non-flav. phenols',
'proanth',
'col. int.', 'col. hue',
'OD ratio'
,'proline']
df.head()
| alcohol | malic acid | ash | ash alkalinity | magnesium | tot. phenols | flavanoids | non-flav. phenols | proanth | col. int. | col. hue | OD ratio | proline | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1.691550 | -0.346811 | 0.487926 | -0.809251 | 0.930918 | 2.491446 | 1.466525 | -0.981875 | 1.032155 | 1.186068 | -0.427544 | 1.184071 | 2.334574 |
| 1 | -0.284874 | 0.981731 | -1.412851 | -1.049479 | -1.386122 | -1.065731 | -0.782361 | 0.549108 | -1.333159 | -0.717240 | -1.129518 | -0.694478 | -1.193845 |
| 2 | 0.209232 | 2.561618 | -0.170035 | 0.752231 | -0.473348 | -0.889474 | -1.404820 | 1.999513 | -0.071658 | 1.229325 | -1.568252 | -1.598441 | -0.069719 |
| 3 | -0.346638 | -0.526343 | -0.316249 | 0.902373 | -1.105268 | -1.466313 | -0.270338 | 0.951998 | 0.068508 | -0.760497 | -0.339797 | -0.270745 | -0.824444 |
| 4 | 0.196879 | 0.021231 | 1.109334 | -0.268738 | 0.088358 | 0.808997 | 1.215533 | -0.498407 | 2.135968 | 0.269020 | 0.318304 | 0.788587 | 1.395148 |
cultivares=['Nebbiolo', 'Barberas', 'Grignolino']
y_cultivar = [ cultivares[int(lbl)-1] for lbl in y_train]
df = pd.DataFrame(y_cultivar)
df.columns = ['Cultivar']
df.head()
| Cultivar | |
|---|---|
| 0 | Nebbiolo |
| 1 | Barberas |
| 2 | Grignolino |
| 3 | Barberas |
| 4 | Nebbiolo |
Ejercicio 1. Clasificación del conjunto de entrenamiento y cálculo de errores#
Utiliza un SOM del mismo tamaño y malla que en la sección anterior y el comando supersom del paquete kohonen para obtener la clasificación del conjunto testdata.
Halla la matriz de confusion y dertermina el error cometido en la clasificación.
nrows = 20
ncols = 20
smm = som(nrows=nrows,ncols=ncols)
nrows = 20
ncols = 20
dimensions = X_train.shape[1]
#vicinity = "rectangular"
vicinity = "hexagonal"
smm = som(nrows=nrows,
ncols=ncols,
dimension=dimensions,
vicinity=vicinity)
smm.train_super_SOM(train_data=X_train, y_train=y_train, epochs=10)
Show code cell output
0%| | 0/10 [00:00<?, ?it/s]
40%|████████████████████████████████████████████████████████▍ | 4/10 [00:00<00:00, 38.14it/s]
90%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████▉ | 9/10 [00:00<00:00, 39.23it/s]
100%|████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████| 10/10 [00:00<00:00, 38.09it/s]
<som.som at 0x1c9c36d5d90>
y_pred = smm.getPredict(X_train)
smm.score(y_pred, y_train)
0.9596774193548387
y_pred = smm.getPredict(X_test)
smm.score(y_pred, y_test)
0.9444444444444444
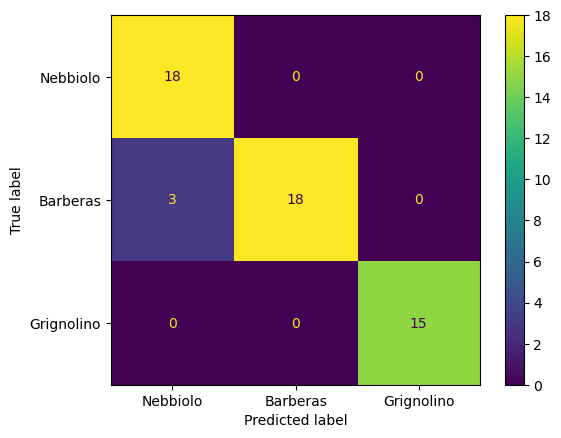
Matriz de confusión#
CMD = ConfusionMatrixDisplay(confusion_matrix(y_true=y_test, y_pred=y_pred), display_labels=cultivares).plot()
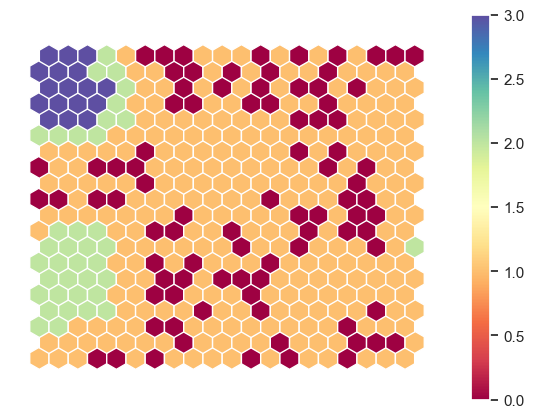
Mapa con las estimaciones#
somutils.plotEstimationMap(pSom=smm, figsize=(7,5))
Ejercicio#
Repite la práctica (clustering y clasificación) usando:
subconjuntos de los datos
wine(nota: scatter matrix)variando los parámetros del SOM (neuronas, funciones) Compara los resultados obtenidos.
